自然科学研究機構 生命創成探究センター/分子科学研究所の奥村久士准教授は谷本勝一特任研究員、伊藤暁助教とともにスーパーコンピューターを使って分子動力学シミュレーション[注1]を行い、薬剤であるレムデシビルやアビガンなどが新型コロナウイルスのRNA依存性RNAポリメラーゼ[注2]に取り込まれる過程を世界で初めて明らかにしました。
本研究成果はアメリカ生物物理学会が発行する国際学術誌Biophysical Journalに2021年8月1日付で掲載されました。
新型コロナウイルスの遺伝子を複製するタンパク質にレムデシビル、アビガンが取り込まれる過程を解明
研究の背景
新型コロナウイルス感染症(COVID-19)に対する治療薬として現在レムデシビルやアビガンが注目されています。これらの薬は新型コロナウイルスのRNA依存性RNAポリメラーゼに作用すると考えられています。RNAポリメラーゼはアデノシン三リン酸(ATP)などのヌクレオチド[注3]を取り込んで新型コロナウイルスの遺伝情報を持つRNAを複製するタンパク質です。レムデシビルやアビガンはATPなどの代わりにRNAポリメラーゼに取り込まれてRNAの複製を阻害すると期待されています。しかし、レムデシビル、アビガン、ATPなどがRNAポリメラーゼにどのように取り込まれるのか分かっていませんでした。
研究の成果
奥村准教授らの研究グループはRNAポリメラーゼの周りにレムデシビル、アビガン、ATPのいずれかを100個配置した3種類の分子動力学シミュレーションを実行しました。これらの分子はいずれもリン酸基を持っており、リン酸基の持つマイナス電荷がRNAポリメラーゼの結合サイト[注4]にあるマグネシウムイオンのプラス電荷に引き付けられて結合することが分かりました。また、これらの薬剤などがRNAポリメラーゼの結合サイトにたどり着くまでの経路は主に3つであることを特定しました。これらの経路のうち2つの経路上ではプラス電荷を持つアミノ酸であるリジン残基が結合サイトに向かって一列に並んでいます。リジン残基がレムデシビルなどのリン酸基を引きよせ、それを隣のリジン残基に受け渡すことを繰り返しながら、バケツリレーのように薬剤などを結合サイトに運んでいる様子を分子動力学シミュレーションにより観測することに成功しました。3つ目の経路は薬剤などがどのアミノ酸残基にも接触せずに直接結合サイトに到達するものでした。これらの結果から、新型コロナウイルスのRNAポリメラーゼは静電的相互作用によってATPやレムデシビルなどを取り込んでいることおよびその効率を高めるためにまるで触手を広げるかのようにリジン残基を結合サイトに向かって一列に並べ、バケツリレーの要領で結合サイトに運んでいることが明らかになりました。
さらに統計誤差の範囲内ではありますが、ATPよりもアビガンの方が、アビガンよりもレムデシビルの方がRNAポリメラーゼの結合サイトに取り込まれる確率が高い傾向にあることが示唆されました。しかし、レムデシビルの方がアビガンより効果が高いと断言できるほどには十分な統計量が得られていないため、明確に判定するにはさらなる計算が必要です。
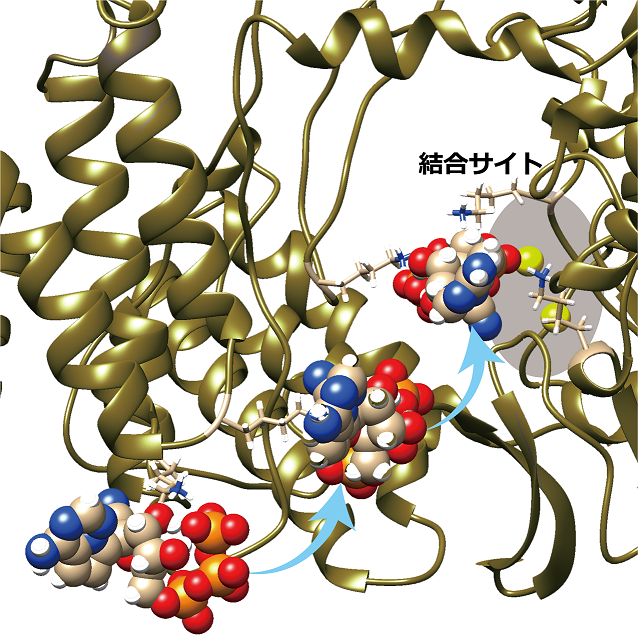
【図】
レムデシビル(球モデルで表示)が複数のリジン残基(棒モデルで表示)に次々と受け渡されながらRNAポリメラーゼ(リボンモデルで表示)の結合サイトにある2個のマグネシウムイオン(黄緑色の球)に運ばれている様子を示す。リジン残基があたかもバケツリレーのようにレムデシビルを運んでいることが分かる。
【動画】
SARS-CoV-2のRNAポリメラーゼの結合サイトにレムデシビルが転送される様子を分子動力学シミュレーションに基づいて作成した動画です。
この研究の社会的意義
この研究により新型コロナウイルスのRNAポリメラーゼがRNAを複製するためにATPなどのヌクレオチドを効率的に取り込むメカニズムが世界で初めて解明されました。また、レムデシビルやアビガンも同様のメカニズムによりRNAポリメラーゼに取り込まれることも明らかになりました。この発見は将来的に新型コロナウイルスの複製を抑制するためのより効果的な薬剤の開発に貢献することが期待されます。さらに、新型コロナウイルスだけでなく重症急性呼吸器症候群(SARS)コロナウイルスのRNAポリメラーゼでも同様にリジン残基が一列に並んでいることが分かっています。したがって、本研究の発見は新型コロナウイルスと同様のRNAポリメラーゼを持つ他のウイルスに対する薬剤開発にも貢献すると考えられます。
用語解説
注1.分子動力学シミュレーション:
原子や分子の配置から力を計算し、ニュートンの運動方程式を数値的に解くことで仮想的に原子や分子の運動をコンピューター上で再現する計算手法。
注2.RNA依存性RNAポリメラーゼ:
RNAを鋳型としてRNAを複製する酵素。
注3.ヌクレオチド:
リン酸、糖、塩基が結合した化合物でDNAおよびRNAの最小構成単位。
注4.結合サイト:
この研究ではRNAポリメラーゼにおいてRNAを複製している所。
掲載論文
雑誌名:Biophysical Journal
論文タイトル:
“Bucket brigade” using lysine residues in RNA-dependent RNA polymerase of SARS-CoV-2
(SARS-CoV-2のRNA依存性RNAポリメラーゼが行うリジン残基による「バケツリレー」)
著者:
谷本 勝一 (自然科学研究機構 分子科学研究所 特任研究員)
伊藤 暁 (自然科学研究機構 分子科学研究所/生命創成探究センター 助教)
奥村 久士 (自然科学研究機構 生命創成探究センター/分子科学研究所 准教授)
掲載日:2021 年8 月1日(オンライン公開)
研究グループ
自然科学研究機構 生命創成探究センター/分子科学研究所 奥村グループ
谷本 勝一、伊藤 暁、奥村 久士
研究サポート
本研究は自然科学研究機構 岡崎共通研究施設計算科学研究センターのスーパーコンピューター高性能分子シミュレーターとHPCIシステム利用研究課題(課題番号:hp200142)を通じて提供された東京工業大学のスーパーコンピューターTSUBAME3.0を利用して実施されました。
本件に関するお問い合わせ先
研究について
自然科学研究機構 生命創成探究センター/分子科学研究所
准教授 奥村 久士(おくむら ひさし)
TEL:0564-59-5213
E-mail:hokumura_at_ims.ac.jp
※_at_は@にご変更ください。
報道について
自然科学研究機構 生命創成探究センター 広報担当
TEL:0564-59-5201 FAX:0564-59-5202
E-mail: press_at_excells.orion.ac.jp
自然科学研究機構 分子科学研究所 研究力強化戦略室 広報担当
TEL:0564-55-7209 FAX:0564-55-7374
E-mail: press_at_ims.ac.jp
※_at_は@にご変更ください。

