名古屋市立大学大学院理学研究科の温琛涛(ウェン・チェンタオ)博士研究員、木村幸太郎教授、米国コロンビア大学のElizabeth Hillman教授、九州大学の石原健教授、自然科学研究機構生命創成探究センター/生理学研究所の根本知己教授、同センター/基礎生物学研究所の青木一洋教授らの国際共同研究グループは、さまざまな先端的顕微鏡によって三次元ビデオとして撮影された100~1000個ほどの細胞を自動的に追跡して細胞活動の解析を可能にする初めての人工知能ソフト「3DeeCellTracker(スリーディー・セル・トラッカー)」を開発しました。
この研究の成果は、基礎生命科学研究や臨床応用研究における画像解析のデジタルトランスフォーメーションに大きく貢献することが期待されます。この論文は、温博士を筆頭著者として国際生命科学専門誌eLifeに3月30日午後5時(英国の午前8時)に発表されます。
三次元ビデオ中の細胞集団を自動的に追跡する世界初の人工知能技術 ~小動物の脳・心臓や三次元培養されたがん細胞集団の活動などが明らかに〜
研究の背景
さまざまな顕微鏡技術が飛躍的に発達したことによって、たくさんの細胞の活動を体の中に近い三次元の状態でビデオ記録することが簡単になってきました(注1)。三次元ビデオ(注2)として撮影された細胞の位置は時間に連れて動いていくので、それぞれの細胞を正しく認識して追跡するソフトが必要です(注3)。しかし三次元ビデオの中の数多くの細胞を追跡するソフトを作ることはとても困難であり、世界中の多くの研究室で「細胞活動の三次元ビデオ」のビッグデータは解析されないままどんどん蓄積していました。
研究の成果
研究グループは、「深層学習」(注4)と呼ばれる人工知能技術を用いることで、三次元ビデオデータ中の数多くの細胞をほぼ自動的に追跡することに世界で初めて成功しました。生命科学研究の画像データの解析では一般的に数多くの細かい調整(パラメータ設定)が必要ですが、本プログラムは深層学習を用いているのでこの調整がすぐに済みます。さらに、深層学習技術が持つ高い柔軟性のために、日本や米国のさまざまな研究室の大きく異なる最先端顕微鏡(注5)によって撮影された細胞活動をわずかな設定の変更によって追跡および解析することができました。具体的には、線虫の脳の神経細胞や熱帯魚ゼブラフィッシュの心筋細胞(共におよそ100個)、そしてガン細胞およそ1000個の三次元集団が解析されました(図1)。
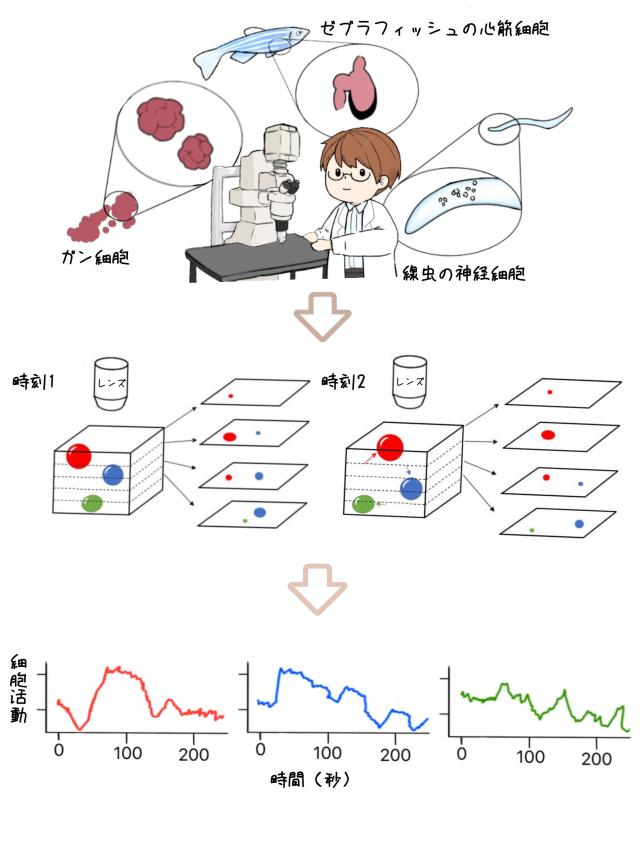
本研究の流れ。さまざまな生物の細胞を最先端の顕微鏡によって観察し(図上)、そこから得られた三次元ビデオの中の細胞を人工知能技術で自動的に追跡し(図中)、それぞれの細胞の活動を解析することが初めて可能になった(図下)。
本研究のプログラムは現在広く使われているPythonというプログラム言語で書かれており、一般に販売されているGPU付きのデスクトップPC上(注6)で動かすことができます。驚くべきことに、過去の研究では計算センターの高性能計算機システム(200コアのPCクラスター)が必要だった複雑な三次元ビデオ画像も、1台のGPU付きデスクトップPCで解析することに成功しました。
研究のポイント
- 三次元ビデオとして撮影された多数の細胞の動きを自動的に追跡することはとても難しかった。
- デスクトップPCで動く深層学習技術を用いた人工知能ソフト「3DeeCellTracker」を開発した。
- 計算センターレベルの性能が必要だった解析も1台のデスクトップPCで可能になった。
- これまでは不可能だった「さまざまな顕微鏡によって撮影された三次元ビデオデータからの細胞活動の解析」が飛躍的に容易になった。
- 線虫の脳全体の神経活動、ゼブラフィッシュの拍動する心臓の細胞活動、三次元のがん細胞集団の活動を、実際に計測・解析した。
研究の意義と今後の展開や社会的意義など
現在、人工知能技術を用いたビッグデータ解析などによって生命科学研究のデジタルトランスフォーメーションをめざす「バイオDX」が注目を集めています。解析されないまま蓄積されている「細胞活動の三次元ビデオ」というビッグデータの自動的かつ大量の解析を可能にする人工知能ソフト3DeeCellTrackerは、小型実験生物などを用いた基礎生命科学研究やガン細胞に対する薬剤開発のような応用研究における画像解析のデジタルトランスフォーメーションに大きく貢献すると期待できます。
用語解説
注1. 細胞の活動の記録
私たちの体は、数十兆個の細胞からできています。それぞれの細胞は、栄養分を取り込んでタンパク質を作るなどとともに、細胞の中のカルシウムやナトリウムなどイオン分子の濃度を積極的に変化させて周囲の細胞に伝えたりしています。ノーベル賞受賞者である下村脩先生が発見された緑色蛍光タンパク質を改良することによって、さまざまな細胞の活動を顕微鏡によって記録できる技術が開発されています。
注2. 三次元ビデオ
私たちがスマホなどで撮影する写真は、「縦・横」(二次元)の画像です。また、この写真撮影を高速で繰り返すことによって、動いているように見える「ビデオ」となります。
顕微鏡の場合はピントが合う「焦点面」を狭く設定(ポートレートの写真のように、特定の距離の対象だけはっきりさせる)した上で、高速で焦点面の高さを変えることで「縦・横・高さ」を持つ三次元の画像を撮ることができます。そして、この三次元画像の取得を繰り返すことで、「三次元ビデオ」を記録することができます。
三次元画像を記録してコンピュータの中で再現すれば、(3Dアクションゲームのように)自由な視点から細胞の様子やその活動を観察することができます。
注3. 細胞の認識と追跡のためのソフト
例えば動いている心臓の100個の細胞の活動を三次元ビデオデータの中から計測するためには、「(1) 細胞とそれ以外の部分を認識する」「(2) それぞれの細胞に1から100の番号を付ける」「(3) 心臓の動きに連れて位置が変わる100個の細胞を追跡する」という処理を自動的に行うことが必要です。
1つめの「認識」は、u-netと呼ばれる人工知能ソフト(深層学習;次項目参照)の誕生によって容易に行うことができるようになりました。また、2つめの「番号付け」も昔からの技術によって簡単に行うことができます。
しかし、3つめの「三次元ビデオデータでの細胞追跡」を行うことができる人工知能ソフトは全く登場していませんでした。最近の人工知能技術の発展を考えればとても不思議なことですが、これには理由がありました。深層学習を用いるためには人が記録した大量の「正解」となるデータ(教師データ)が必要なのですが、実際に記録された三次元ビデオデータの中での細胞の位置を正確かつ大量に記録することはほぼ不可能だったのです。本研究チームはこの問題を、「三次元ビデオデータ中での細胞の動きをまねた人工的なデータ」を作成して深層学習の教師データとして用いることによって解決しました。
注4. 深層学習
人工知能技術の1つ。人の脳の構造をまねて、人工的な神経細胞をネットワーク上に結びつけることで、1つ1つの人工神経細胞が行う計算処理自体は単純でも、ネットワーク全体では従来のプログラムでは不可能だったレベルの計算処理が可能になりました。
注5. 三次元ビデオ記録用のさまざまな最先端顕微鏡
注2でも説明したように、三次元ビデオを撮るためには、顕微鏡で観察する画像の焦点面を非常に狭く絞った上で、素早く正確にほんのわずかな距離だけ(本研究では1ミリメートルの1/1000程度)移動させて撮影を繰り返す必要があります。今回の論文では、「スピニングディスク型共焦点顕微鏡」「掃引型共焦点平面励起顕微鏡 (SCAPE 2.0; Voleti etl al., Nat Meth 2019)」「二光子励起顕微鏡」という、3つの全く異なる最先端の顕微鏡が用いられました。
注6. GPU付きデスクトップPC
GPU (Graphics processing unit) とは画像処理のためのパソコンのパーツです。元々はビデオゲームなどでの画面処理を高速化するために導入されていましたが、比較的単純な計算を大量に行う必要がある深層学習などの計算処理にも適していることが分かり、広く用いられています。
研究助成
本研究は、新学術領域研究「生物移動情報学」(木村幸太郎、JP16H06545)、科学研究費基盤研究S(木村幸太郎、20H05700)、米国国立神経疾患・脳卒中研究所(Elizabeth Hillman、U01NS094296 and UF1NS108213)、米国国立がん研究所(Elizabeth Hillman、U01CA236554)、新学術領域研究「脳情報動態」(石原健、JP18H05135)、NTT-九州大学基礎科学共同研究プログラム(石原健)、文部科学省博士課程教育リーディングプログラム「生態統御ネットワーク医学教育プログラム」(三浦拓也)、名古屋市立大学特別研究奨励費(木村幸太郎、48、1912011、1921102)、自然科学研究機構分野融合型共同研究事業(木村幸太郎、01112002)、豊秋財団研究費助成(木村幸太郎)、理化学研究所革新知能統合研究センター共同研究(木村幸太郎)の支援を受けて行われました。
論文情報
論文タイトル:
“3DeeCellTracker, a deep learning-based pipeline for segmenting and tracking cells in 3D time lapse images”
(三次元時系列画像における細胞の分離と追跡のための深層学習によるパイプライン、3DeeCellTraker)
著者:
Chentao Wen*, Takuya Miura, Venkatakaushik Voleti, Kazushi Yamaguchi, Motosuke Tsutsumi, Kei Yamamoto, Kohei Otomo, Yukako Fujie, Takayuki Teramoto, Takeshi Ishihara, Kazuhiro Aoki, Tomomi Nemoto, Elizabeth M.C. Hillman, Koutarou D. Kimura
(*はcorresponding author)
所属:
温琛涛(名古屋市立大学大学院理学研究科、博士研究員、本論文の責任著者)
三浦拓也(大阪大学大学院理学研究科生物科学専攻、大学院生)(当時)
Venkatakaushik Voleti(米国コロンビア大学医用工学および放射線学科、ザッカーマン心脳行動研究所、大学院生)(当時)
山口和志(北海道大学大学院情報科学研究院、自然科学研究機構生理学研究所、大学院生)
堤元佐(自然科学研究機構生理学研究所/生命創成探究センター、特任助教)
山本啓(自然科学研究機構基礎生物学研究所、総合研究大学院大学、大学院生)
大友康平(自然科学研究機構生理学研究所/生命創成探究センター、総合研究大学院大学、助教)
藤江由香子(大阪大学大学院理学研究科生物科学専攻、技術補佐員)
寺本孝行(九州大学大学院理学研究院、准教授)
石原健(九州大学大学院理学研究院、教授)
青木一洋(自然科学研究機構基礎生物学研究所/生命創成探究センター、総合研究大学院大学、教授)
根本知己(自然科学研究機構生理学研究所/生命創成探究センター、総合研究大学院大学、教授)
Elizabeth M.C. Hillman(米国コロンビア大学医用工学および放射線学科、ザッカーマン心脳行動研究所、教授)
木村幸太郎(名古屋市立大学大学院理学研究科、教授;大阪大学大学院理学研究科、招へい教授;理化学研究所革新知能統合研究センター、客員研究員)
掲載誌:eLife
本件に関するお問い合わせ先
研究に関するお問い合わせ
名古屋市立大学 大学院理学研究科
教授 木村 幸太郎
TEL: 052-872-5016 FAX:052-872-1531(事務室)
E-mail:kokimura_at_nsc.nagoya-cu.ac.jp
※_at_は@にご変更ください。
報道に関するお問い合わせ
名古屋市立大学 山の畑事務室
名古屋市瑞穂区瑞穂町字山の畑1 滝子キャンパス
TEL:052-872-3461 FAX:052-872-1531
E-mail: kanri-jimu_at_sec.nagoya-cu.ac.jp
※_at_は@にご変更ください。

